多目的最適化
目次
はじめに
最近たまたま多目的最適化を扱う機会があり、DEAPを使って最適化を実施したので、その時のメモです。
DEAPのチュートリアルに、GAでの多目的最適化の例があったので使わせてもらいました。
パレート最適解
多目的最適化の場合、一般的に最適解は1つに定まりません。
例えば2つの目的関数を、どちらもできるだけ小さくしたい場合を考えてみます。
1つ目の目的関数が最小値を取ったとしても、同じ設計変数で、もう一つの目的関数が最小値を取るとは限りません。
GAでの多目的最適化では、最終世代の個体の適応度(目的関数)をグラフにプロットしてやると、ある曲線上に乗ってきます。
この曲線のことをパレートライン(又はパレートフロント)と呼び、目的関数がパレートライン上に乗る解はパレート最適解と呼ばれています。
多目的最適化では、パレートラインが確認できるまで最適化ループを回してやるのが一般的で、パレートラインを得る事が最終目標と言ってもいいです。
パレートライン上の解は全て最適解で無数にあるので、後は適当に好きなものを選ぶ形になります。
多目的最適化の選択アルゴリズム
多目的最適化では、パレートライン上の最適解集合を如何に広域まで求めるかという事が重要になってくるので、選択アルゴリズムを工夫する必要があります。
選択アルゴリズムは色々あるみたいですが、NSGA-Ⅱ(Non-dominated S orting Genetic A lgorithms-II)というのが一般的みたいです。
ネットで調べてみたところ、どなたかの修士論文が検索に引っかかり、NSGA-Ⅱのアルゴリズムが詳しく書かれてありました。
チョット読んでみましたが、まあ先人の知恵と言いましょうか、中々に難しいです。複雑なので詳細は理解できませんでしたが、ざっくり言うと、パレートフロントに近い個体をランク付けして、ランクが高い個体を優先的に次世代へ残して行く感じです。
とりあえず今回の目的は、DEAPのチュートリアル内容が理解できて、多目的最適化を実施できることとして、進めて行きます。
サンプルコード
GitHub上のDEAPのexamplesフォルダ内に、NSGA-Ⅱを使った多目的最適化のサンプルスクリプトexamples/ga/nsga2.pyが用意されています。
以下のコードは、上記のサンプルスクリプトと基本的には同じものですが、初期世代と最終世代で、パレートラインがどのように変化するかを見たかったので、各個体の適応度をプロットしたグラフを出力する記述を追加しました。
また、長くなるのでコメントは削除してあります。
ちなみに、現時点での話ですがpipでDEAPをインストールした場合は、バージョンが古いようでこのコードを実行するとエラーが出ます。最新版をGitHubからとってきてインストールし直す必要があるので注意してください。
コードの解説
creator.create("FitnessMin", base.Fitness, weights=(-1.0, -1.0))
目的関数が2つあり、どちらも最小化する設定です。
BOUND_LOW, BOUND_UP = 0.0, 1.0
遺伝子が取り得る値の範囲を指定しています。遺伝子は0.0~1.0の間の連続値をとります。
NDIM = 30
1つの個体内の遺伝子の数を指定しています。遺伝子の数は30個です。
def uniform(low, up, size=None): try: return [random.uniform(a, b) for a, b in zip(low, up)] except TypeError: return [random.uniform(a, b) for a, b in zip([low] * size, [up] * size)]
遺伝子生成の関数です。うまいですね。遺伝子の取り得る範囲を指定するlowとupが、listでも指定できるようになってます。
listにした場合は、各遺伝子ごとに取り得る範囲を指定できますね。
toolbox.register("evaluate", benchmarks.zdt1)
適応度を評価する目的関数の指定です。deap/benchmarks/__init__.pyに定義があります。
以下zdt1の定義部です。
def zdt1(individual): """ZDT1 multiobjective function. :math:`g(\\mathbf{x}) = 1 + \\frac{9}{n-1}\\sum_{i=2}^n x_i` :math:`f_{\\text{ZDT1}1}(\\mathbf{x}) = x_1` :math:`f_{\\text{ZDT1}2}(\\mathbf{x}) = g(\\mathbf{x})\\left[1 - \\sqrt{\\frac{x_1}{g(\\mathbf{x})}}\\right]` """ g = 1.0 + 9.0*sum(individual[1:])/(len(individual)-1) f1 = individual[0] f2 = g * (1 - sqrt(f1/g)) return f1, f2
何でしょうねこれ? 取りあえず個体individualを引数にして、2つの目的関数値f1, f2を出力していますね。多目的最適化のベンチマークでよく使われる関数の様です。
toolbox.register("select", tools.selNSGA2)
選択アルゴリズムにNSGA-Ⅱを使った選択関数tools.selNSGA2を指定しています。定義はdeap/tools/emo.pyの一番最初に書かれていますが、関数が入れ子になってて何をやっているかよくわかりませんでした。
NGEN = 250 MU = 100 CXPB = 0.9
NGEN = 250が世代ループの回数で、MU = 100は世代内の個体数、CXPB = 0.9は交叉率です。交叉率随分高い設定ですね。
stats = tools.Statistics(lambda ind: ind.fitness.values) stats.register("min", numpy.min, axis=0) stats.register("max", numpy.max, axis=0) logbook = tools.Logbook() logbook.header = "gen", "evals", "min", "max"
世代ループ中のログに何を出力するかの設定です。世代内の個体の適応度のminとmaxを各適応度別で出力します。
record = stats.compile(pop) logbook.record(gen=0, evals=len(invalid_ind), **record) print(logbook.stream)
ログ出力の為に、適応度のminとmaxを算出する記述です。まあ、これはあまり触れないでおきましょう。
for ind1, ind2 in zip(offspring[::2], offspring[1::2]): if random.random() <= CXPB: toolbox.mate(ind1, ind2) toolbox.mutate(ind1) toolbox.mutate(ind2) del ind1.fitness.values, ind2.fitness.values
交叉と突然変異です。一気にやっちゃってます。
print("Final population hypervolume is %f" % hypervolume(pop, [11.0, 11.0]))
最終世代のハイパーボリュームを出力する記述です。
ハイパーボリュームというのは、適応度空間上のボリュームで、ある参照点(上述では[11.0, 11.0])と各個体の適応度が作る超空間上の立方体を世代内の全個体で作成し、その和集合の体積を算出したものです。(超空間上の立方体の和集合の体積ってどうやって計算するんですかね。全く思いつかない。。)
今回の場合は、個体内の適応度は2こあるので、適応度空間は2次元になり、ハイパーボリュームは所謂面積になります。
ハイパーボリュームの値が大きいほど、パレートラインが広範囲に広がっていることを表し、良いパレートラインが得られていることになります。
参照点は好きな値を指定してOKです。評価する目的間数の数と同じだけ指定します。
スクリプトの実行結果
上記スクリプトの実行ログです。 今回の例では2つある目的関数をどちらも最小化するのでminの値に注目します。
gen evals min max 0 100 [ 0.00776997 2.62064288] [ 0.99151989 5.4725374 ] 1 100 [ 0.00776997 2.36779157] [ 0.99151989 5.76456845] 2 100 [ 0.00407038 2.34057121] [ 0.929251 5.96207363] 3 100 [ 0.00275913 2.13467352] [ 0.96942078 5.27230708] ... ... 246 100 [ 7.26551516e-08 7.70375954e-04] [ 0.99996838 1.00580077] 247 100 [ 7.26551516e-08 7.41530716e-04] [ 0.99999967 1.00580077] 248 100 [ 7.26551516e-08 7.41530716e-04] [ 0.99999967 1.00580077] 249 100 [ 7.26551516e-08 7.41530716e-04] [ 0.99999967 1.00580077] Final population hypervolume is 120.651895
最終世代ではmin値は[ 7.26551516e-08 7.41530716e-04]まで落ちてますね。
ただ、あくまでこれは適応度ごとにmin値をとった物です。2つある適応度のmin値[ 7.26551516e-08 7.41530716e-04]を1つの個体が同時にとっているわけでは無いので注意してください。
以下は、初期世代と最終世代で、各個体の2つある適応度の内、1つ目の適応度をf1、2つ目の適応度をf2として、グラフにプロットしたものです。
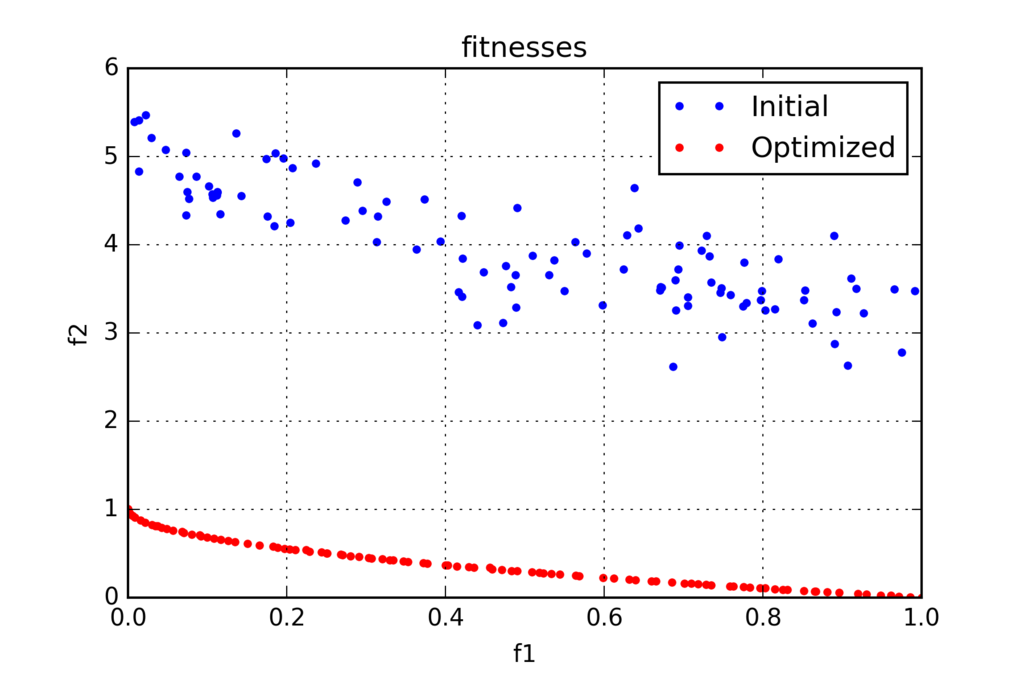
青い点が初期世代、赤い点が最終世代です。最終世代ではずいぶん綺麗なパレートラインが得られてますね。